
Reihenfolge
der Auswerteschritte :
1.
Aufstellen der MPR-3D mit MRIcro ![]()
2.
Umbenennen der EPI mit TOTAL-Commander
![]()
3.
Nullpunkt bestimmen der MPR-3D
4.
Nullpunkt bestimmen und Aufstellen der EPI
5.
MPR-3D in die EPI-Ordner kopieren
8.
Coregistrierung der MPR-3D mit EPI
11.
Smoothen der EPI
1) Anatomische Dateien
(MPR-3D) richtig orientieren:
Anatomische Datei in MRIcro öffnen,
unter Save-as mit folgenden Optionen abspeichern:
sagittal-left
& flip top-bottom. Save-as erzeugt eine neue Datei, deren Namen mit
x beginnt. Alle weiteren Arbeiten an den MPR-3D bitte nur an diesen richtig
orientierten Dateien (xs-*.img/hdr)
ausführen!
Bilder mit MRIcro
öffnen:
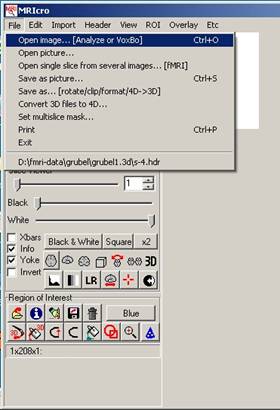
File -> Open Image -> s-*.hdr auswählen
![]()
Speichern:
File -> Save as -> FENSTER:
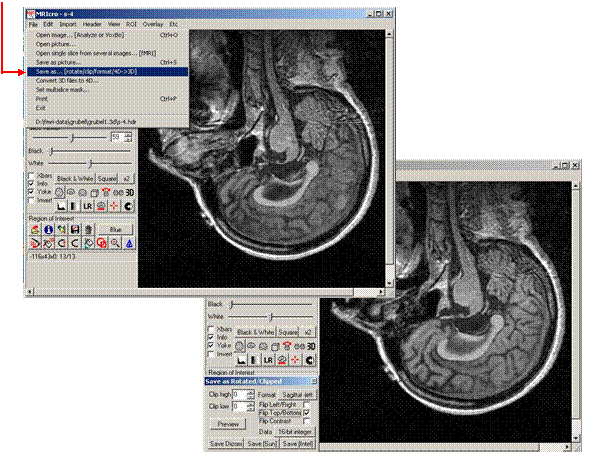
![]() Save as
Rotated/Clipped
Save as
Rotated/Clipped
![]()
![]()
![]()
Format: sagital -left; Flip
Top/Bottom -> Save[Intel] als xs-*.hdr
2) EPI umbenennen:
s-12.img/.hdr muss zu
s-0012.img/.hdr usw. werden. Das ist sehr wichtig, da
sonst SPM die Dateien in der falschen Reihenfolge einliest.
Total Commander öffnen: ![]()
Umzubenennende
Datensätze öffnen
ACHTUNG: das Umbenennen muss für jede Datenart
getrennt durchgeführt werden, d.h. einmal für die .img
einmal für die .hdr:
![]() deshalb nach Erweiterung sortieren (dadurch erhält man die Trennung der .img und .hdr)
deshalb nach Erweiterung sortieren (dadurch erhält man die Trennung der .img und .hdr)
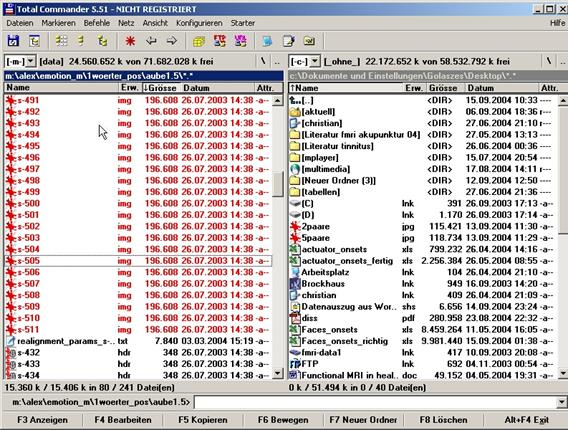
alle s-*.img
markieren (mit SHIFT bzw. STRG)
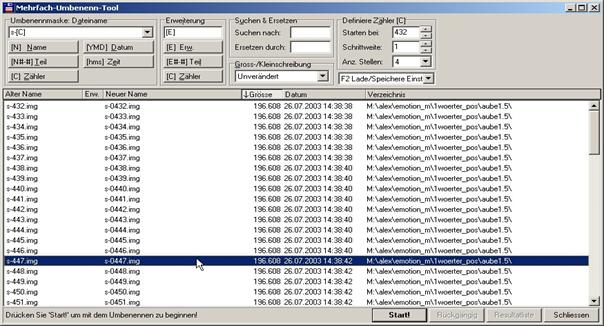
Dateien à Mehrfach-Umbenenn-Tool:
 Dateinname: s-[C] Erweiterung [E] Start bei: erste Nummer des alten Namens
Dateinname: s-[C] Erweiterung [E] Start bei: erste Nummer des alten Namens
 Anz.
Stellen: 4
Anz.
Stellen: 4

![]()
![]()
![]()
Das Ganze danach für die .hdr
wiederholen.
Eventuell vorhandene s-*.mat files
löschen.
Es dürfen nur s-*.img
und s-*.hdr sowie ein Porbanden-log-file
vorhanden sein.
Alle
anderen Files löschen!
SPM99 starten
Matlab starten ![]()
EDU>> spm eingeben
FMRI time-serie anklicken

Maske
erscheint
Startbildschirm
mit Hauptmenü-Maske erscheint
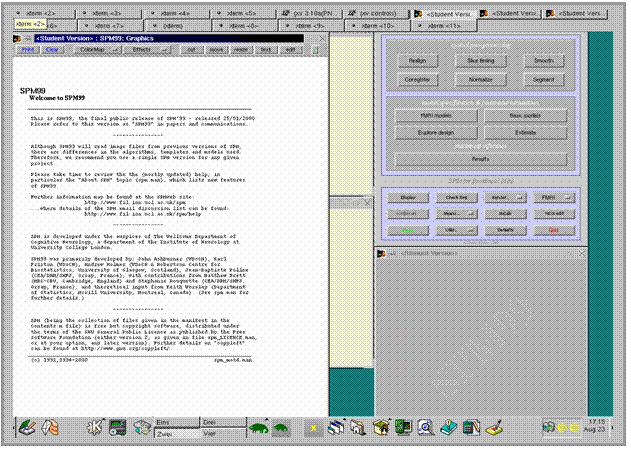
Von hier aus werden alle
Programmfunktionen durch anklicken angewählt und ausgeführt.
 3. Nullpunktfestlegung der MPR-3D
3. Nullpunktfestlegung der MPR-3D
Display anklicken
neues
Fenster öffnet sich:
·
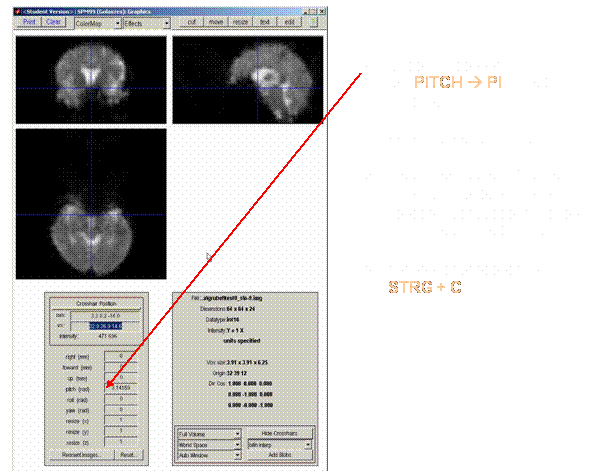
Mit
der Maus das Faden-Kreuz auf die vordere Commissur
klicken ·
VX
Wert markieren mit STRG + C ·
durch Anklicken Vergrößern der D
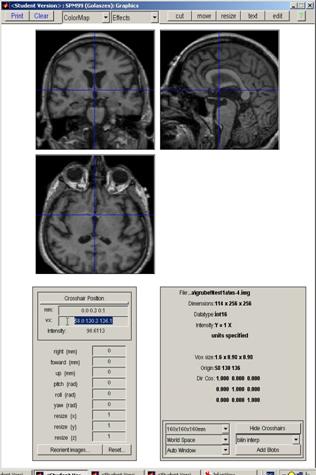
Bilder möglich

HDR-Edit anklicken
![]()
Fenster öffnet sich: Options...
Set
Origion –x,y
& z coordinated of voxel anklicken
Fenster öffnet sich: Origion (x y z)
mit
STRG + V den zuvor markierten VX-Wert einfügen
Fenster öffnet sich: Options...
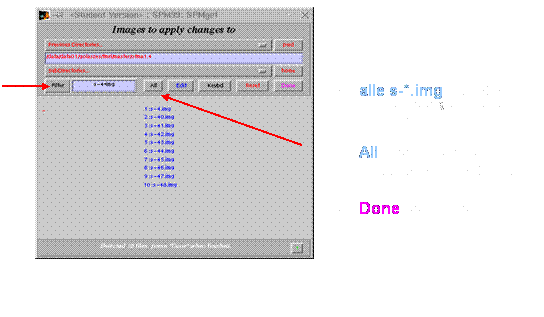
Apply to images anklicken
·
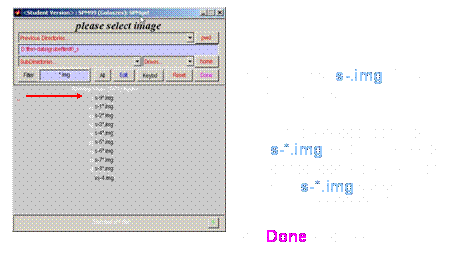
xs-*.img
anklicken
Fenster öffnet sich:
![]()
Fenster öffnet sich: Options...
QUIT anklicken
·
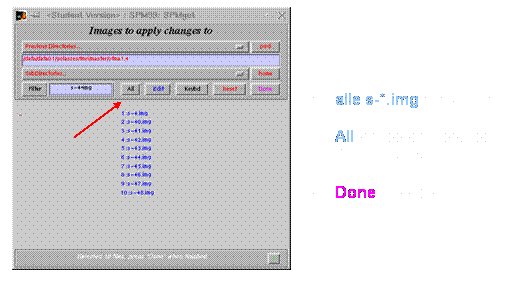
Reorient images
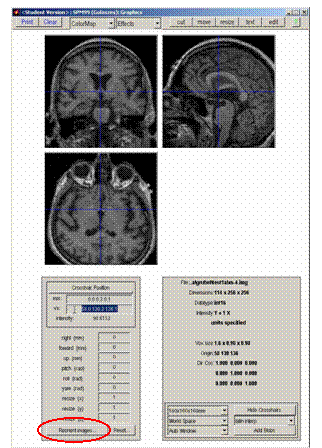
anklicken
·
xs-*.img
anklicken
Fenster öffnet sich:
![]()
 4. Aufstellen und
Nullpunktfestlegung der EPI-Daten
4. Aufstellen und
Nullpunktfestlegung der EPI-Daten
Display anklicken
neues
Fenster öffnet sich:

HDR-Edit anklicken
![]()
Fenster öffnet sich: Options...
Set
Origion –x,y
& z coordinated of voxel anklicken
Fenster öffnet sich: Origion (x y z)
mit
STRG + V den zuvor markierten VX-Wert einfügen
Fenster öffnet sich: Options...
Apply to images anklicken
Fenster öffnet sich:
Fenster öffnet sich: Options...
QUIT anklicken
·
Reorient images
anklicken
Fenster öffnet sich:
5. MPR-3D kopieren
Die zuvor aufgestellten
und mit dem Nullpunkt versehenen MPR-3D (xs-*.img/.hdr) Datensätze müssen
nun in die Verzeichnisse der jeweils zugehörigen EPI-Datensäten
kopiert werden. Am Ende müssen in jedem EPI-Verzeichnis
die entsprechenden MPR-3D Files vorliegen. Die MPR-3D Files sollten kopiert und
nicht verschoben werden. Die separaten 3D-Ordner sollten zur Sicherheit
erhalten bleiben.
Diese Arbeit ist am
Besten mit dem Total Commander ![]() durchzuführen.
durchzuführen.
6. Realignment der EPI-Daten


![]() Realign anklicken
Realign anklicken
Fenster öffnet sich:
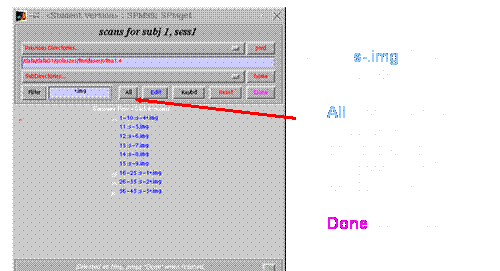
Number of subjects: 1 (Anzahl der Datensätze)
Sessions for subjects: 1 eingeben (immer)
Fenster
öffnet sich:
Wenn
mehrere Datensätze auf einmal angesetzt werden sollen, muss die Anzahl bei Number of subjects eingeben werden.
Danach
öffnet sich für jeden Datensatz das obige Fenster erneut.
Fenster
öffnet sich:
Which
option? Coregister
& Reslice
Reslice Interpol. Method? Sinc interpolation
Create
what? All Images + Mean Image
Adjust
sampling errors? no
|
Säule erscheint – warten |
Realing
erzeugt einen neuen Satz Dateien: rs-*.img
7. Slice Timing der
EPI-Daten
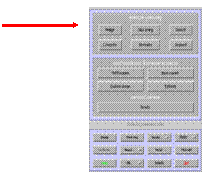
Slice timing anklicken
Fenster öffnet sich:
Number of subjects: 1 (Anzahl der Datensätze)
Fenster
öffnet sich:
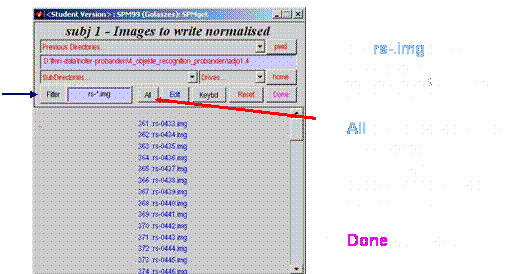
Fenster
öffnet sich:
Select sequence type: descending
Reference Slice: 12
Interscan Interval TR: 3.090
Acquisition Time TA: 1.9583
|
Säule erscheint –
warten |
Erklärung zu den Parametern:
Descending aus folgendem Grund:
Das
erste gemessene Slice in dem Scanner für diese Studie
(Siemens Vision, Radiologie II, Universität Innsbruck) mit der Standard-EPI-Sequenz
ist eigentlich das Unterste vom Gehirn. Also sollte man hier, wären die Bilder
richtig orientiert, ascending eingeben. Aber die
Bilder stehen noch immer real auf dem Kopf! Denn die Motion correction
hat die .mat
Dateien vom Aufstellen ignoriert. Und slice timing ignoriert grundsätzlich alle Info in .mat-Dateien!
Das TR wird vom Scanner (in diesem Beispiel) mit 3s angegeben,
tatsächlich ergibt die Info in den Presentation-Logs
ein TR von 3,090s. Dieser Wert ist genauer.
TA ist die Zeit, wann mit der
Messung der letzten Schicht begonnen wurde. Hier war das TR zwar 3,090 s, aber
die 24 Schichten wurden in 2s gemessen, danach war 1 s Pause. Also wurde die
letzte Schicht gemessen: 2 – (1/24)=1,9583.
Slice timing erzeugt einen neuen Satz Dateien: ars-*.img
8. Coregistrierung der MPR-3D mit der EPI-Daten
![]()
![]() Defaults
anklicken
Defaults
anklicken
Fenster
öffnet sich: Defaults Area ?
Coregistration anklicken
Fenster
öffnet sich: Use
Mutal Information Registration ?
Use Mutal
Information Registration anklicken
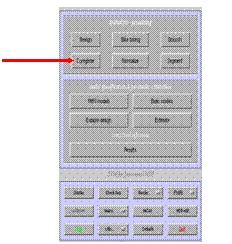
Coregister
anklicken
Number of subjects: 1
(Anzahl der
Datensätze)
Which option: Coregister only
Fenster
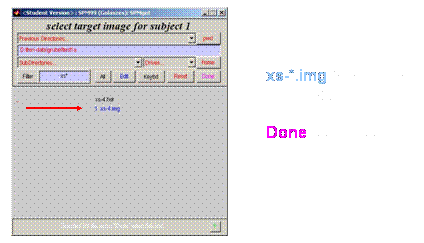
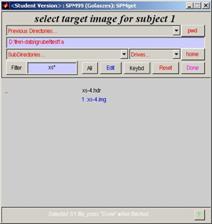
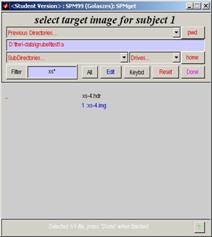
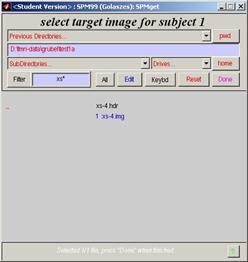
öffnet sich : select target images for subject 1
·
erstes ars.-img File des entsprechenden funktionellen Datensatzes
anklicken
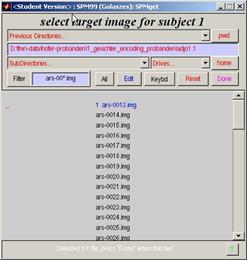
Fenster
öffnet sich: select object images for subject 1
·
xs-4.img (MPR-3D) dieses Datensatzes anklicken ·
Done anklicken
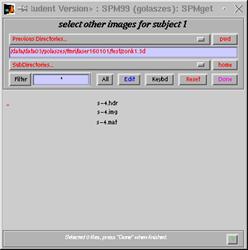
Fenster
öffnet sich: select other images for subject 1
·
nur Done anklicken (nichts weiter
anklicken)
Die Güte der Coregistrierung unbedingt
kontrollieren, davon hängt die Güte der Normalisierung ab! Falls Fehlschlag,
die Defaults umstellen auf „Use default Registration“ und
noch mal laufen lassen. Die Coregistrierung läuft
dann identisch ab, man muss nur vorher noch folgende 2 Einstellungen treffen:
Modality
of first target images ? : target-EPI
Modality
of first object images ? : object-T1 MRI
Kontrollieren: Den Button Check_Reg im SPM Hauptmenü anklicken. Zwei Bilder gleichzeitig
auswählen, ein EPI (ars-000x.img) und das MPR-3D. Mit dem Cursor auf das Bild
klicken und checken, ob die Gehirngrenzen / Corpus Callosum
& Ventrikel von MPR 3D & EPI übereinstimmen.
Normalerweise funktioniert „Use Mutual
Information Registration“ sehr gut, wenn nicht, wie beschrieben
die Defaults ändern und noch einmal probieren.
Coregister erzeugt
keinen neuen Satz Dateien, die Informationen werden in die .mat
files des MPR-3D geschrieben.
9. Normalize der MPR-3D

Defaults anklicken
Fenster öffnet sich: Defaults Area ?
 Spatial Normalisation
anklicken
Spatial Normalisation
anklicken
Fenster öffnet sich: Defaults for
?
Defaults
for Writing Normalized anklicken
Fenster öffnet sich: Bounding Box ?
78:78
–112:76 –50:85 (Default ) anklicken
Fenster öffnet sich: Voxel Sizes ?
Customise anklicken
Fenster öffnet sich: Voxel Sizes 2
2 2
mit
Leerzeichen eingeben!
Defaults anklicken
Fenster öffnet sich: Defaults Area ?
Spatial Normalisation anklicken
Fenster öffnet
sich: Defaults for
?
Defaults
for Parameter Estimation anklicken
Fenster öffnet sich: Affine Starting Estimate ?
Radiological
Convention (L is R) anklicken
Fenster öffnet sich: Allow customised normalisation ?
Disallow Customised anklicken
Fenster öffnet sich: Nonlinear Basics Function ?
7x8x7
anklicken
Fenster öffnet sich: Nonlinear iterations ?
12
nonlinear iteration anklicken
Fenster öffnet sich: Nonlinear Regularization ?
Medium regularization anklicken
Fenster öffnet sich: Mask brain when registering ?
Default
Brain Mask anklicken
Fenster öffnet sich: Mask object brain when
registering ?
Dont Mask object anklicken

![]() Normalize anklicken
Normalize anklicken
Which
option: * Determine Paramerters
& write normalised
subjects : 1 (Anzahl der Datensätze)
Fenster öffnet sich: image to determine
paramerters from
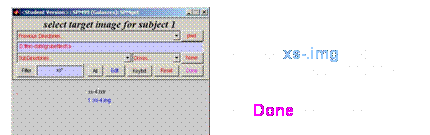
Fenster öffnet sich: image to write
normalised
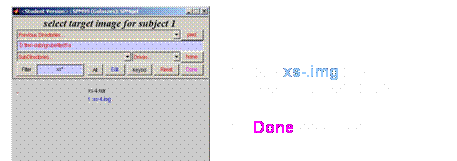
Fenster öffnet sich: template
images
·
T1.img anklicken
·
Done anklicken
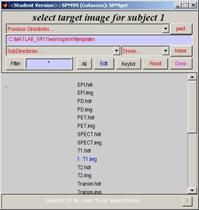
Interpolation
Method: bilinear Interpolation
Normalize der MPR-3D
erzeugt einen neuen Satz Dateien: nxs-*.img
10. Normalize der
EPI-Daten

Defaults anklicken
Fenster öffnet sich: Defaults Area ?
 Spatial Normalisation
anklicken
Spatial Normalisation
anklicken
Fenster öffnet sich: Defaults for
?
Defaults
for Writing Normalized anklicken
Fenster öffnet sich: Bounding Box ?
78:78
–112:76 –50:85 (Default ) anklicken
Fenster öffnet sich: Voxel Sizes ?
Customise anklicken
Fenster öffnet sich: Voxel Sizes 4
4 4
mit
Leerzeichen eingeben!
Defaults anklicken
Fenster öffnet sich: Defaults Area ?
Spatial Normalisation anklicken
Fenster öffnet
sich: Defaults for
?
Defaults
for Parameter Estimation anklicken
Fenster öffnet sich: Affine Starting Estimate ?
Radiological
Convention (L is R) anklicken
Fenster öffnet sich: Allow customised normalisation ?
Disallow Customised anklicken
Fenster öffnet sich: Nonlinear Basics Function ?
7x8x7 anklicken
Fenster öffnet sich: Nonlinear iterations ?
12
nonlinear iteration anklicken
Fenster öffnet sich: Nonlinear Regularization ?
Medium regularization anklicken
Fenster öffnet sich: Mask brain when registering ?
Default
Brain Mask anklicken
Fenster öffnet sich: Mask object brain when
registering ?
Dont Mask object anklicken

![]() Normalize anklicken
Normalize anklicken
Which option: Write normalised only
(Determine
Paramerters
& write normalized)
subjects: 1 (Anzahl der Datensätze)
Fenster öffnet sich: image to determine paramerters
from

Fenster öffnet sich: image to write normalised

Fenster öffnet sich: template
images
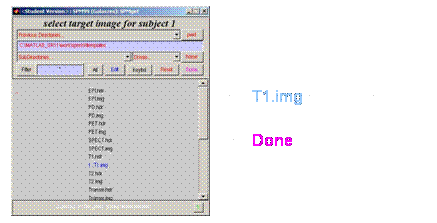
Interpolation
Method: bilinear Interpolation
Normalize der EPI-Daten
erzeugt einen neuen Satz Dateien: nars-*.img

 11. Smoothing der EPI-Daten
11. Smoothing der EPI-Daten
Smooth anklicken
Smoothing (FWH. in
mm) 8 8 8 eingeben
(2-3facher Voxelwert)
mit Leerzeichen eingeben!
Fenster öffnet
sich:
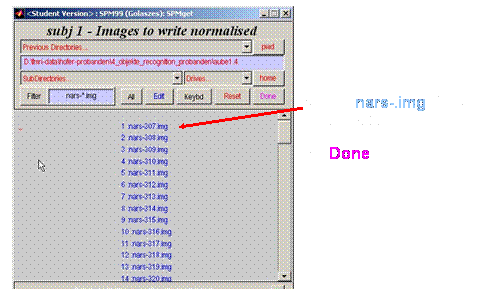
·
Wenn mehrere Datensätze
auf einmal angesetzt werden sollen, nacheinander die entsprechenden nars-.img anwählen ohne jedesmal Done
zu drücken. Done wird erst am Schluss – nach der Anwahl
des letzten Datensatzes - gedrückt.
|
Säule erscheint –
warten |
Smoothen der EPI-Daten
erzeugt einen neuen Satz Dateien: snars-*.img